SimLVSeg With Mamba 实验
1. 主体框架-SimLVSeg
SimLVSeg使基于视频的网络能够从稀疏注释的超声心动图视频中进行一致的LV分割。SimLVSeg包括具有时间掩蔽的自监督预训练,然后是为从稀疏注释中进行LV分割而定制的弱监督学习。我们展示了SimLVSeg如何在最大的2D+时间超声心动图数据集(EchoNet-Dynamics)上实现93.32%(95%CI 93.21-93.43%)的DICE评分,同时更高效,从而优于最先进的解决方案。SimLVSeg兼容两种类型的视频分割网络:2D超级图像和3D分割。为了展示我们方法的有效性,我们提供了广泛的消融研究,包括预训练设置和各种深度学习主干。我们进一步进行了分布外测试,以展示SimLVSeg在未见分布(CAMUS数据集)上的推广能力。该代码可在https://github.com/fadamsyah/SimLVSeg上公开获取。
SimLVSeg使基于视频的网络能够进行LV分段,从而增强性能和更高的时间一致性。SimLVSeg由两个训练阶段组成:具有时间掩蔽的自我监督预训练和LV分割的弱监督学习,专门设计用于解决稀疏注释(标记)超声心动图视频的挑战。
结构
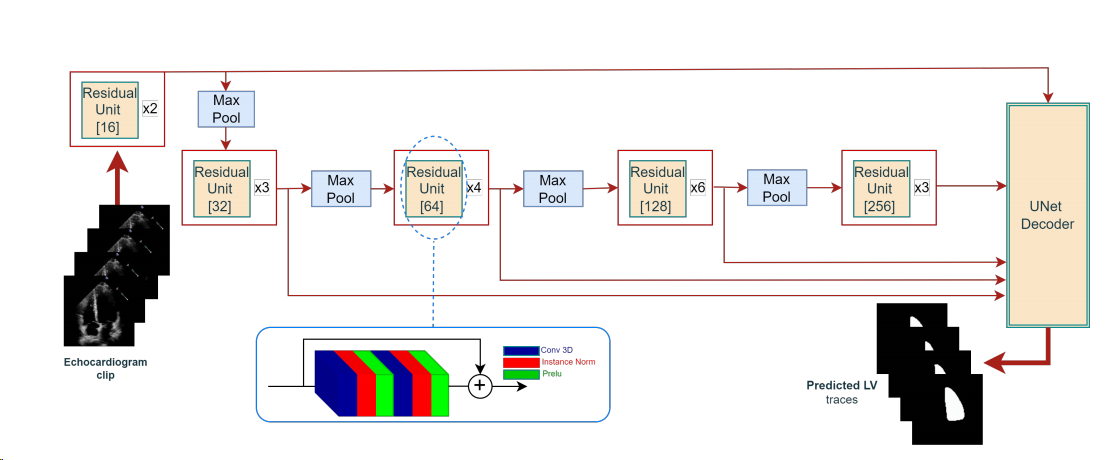
超声心动图视频由堆叠的2D图像组成。将时间轴视为第三维允许3D模型在超声心动图剪辑上分割LV。因此,3D U-Net被用作架构。如图4所示,我们使用具有残余单元的CNN 作为编码器,其具有5个级,其中级输出被传递到解码器。剩余单元包括两个Conv 2D层、两个实例规范层、两个PReLU激活函数和一个跳过连接。
数据集
EchoNet-Dynamics是最大的公开2D+Time心脏超声心动图,其显示了人体心脏的顶部四腔视图。提供了约10,030个心脏超声心动图视频,固定帧大小为112 x 112。
视频长度从28帧到1002帧不等,涵盖多个心跳周期,但只有两个被注释(ED和ES帧)。图1中给出了示例超声心动图序列
实验
作者同时还做了多个消融实验,验证不同的主干网络对该框架性能的影响,实验数据如下:
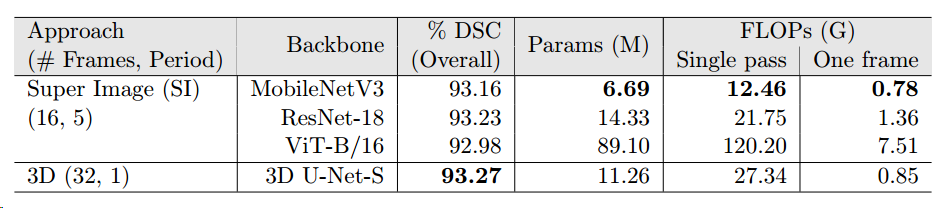
如表2所示,实验表明性能对编码器主干是稳健的。
1 | |
2. 心脏分割模型
2.1 模型修改-SegMamba
SegMamba也是一个分析3D超声心动图的网路,不同的是原论文中实验采用的体积数据,而不是2D图像数据加时间。因此,将SegMamba移植过来作为主干网络时,将时间作为第三个轴,即输入的帧数作为高,SegMamba在原论文实验中表现同样十分优异。详细的集成后代码可以在这里找到https://github.com/52HZMercury/SimLVSeg-with-SegMamba
使用的环境
py3.8 torch 1.12
首先需要注意的就是原论文中,SegMamba第一层是使用的4个通道,这里需要初始化为3个通道进行输入,这里解决之后,多半会遇到tensor对不上的问题,接着往下看
这里tensor对不上的主要原因就是EchoNet-Dynamics数据集是每帧112112的,而SegMamba在原论文中是长宽高为128 * 128 * 128的输入。解决的方法就是把util代码里面的读取视频的代码输出时resize一下,改为128128,至于还有一个128,在SimLVSeg中输入时将帧改为128即可,即对应的高。
1 | |
在经历上面的操作后,在实验时,可能会遇到爆显存的错误(3090),这里的解决方法是,改小精度,将原来的32位改为了16位
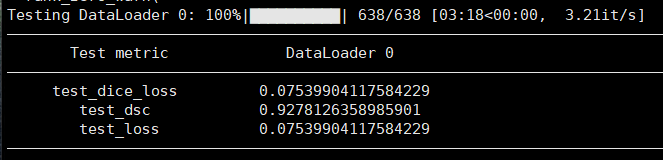
实验结果
Table.2. DSC(Dice Similarity Coefficient)
| Step | DSC | loss |
|---|---|---|
| 2235 | 0.863 | 0.138 |
| 2980 | 0.922 | 0.079 |
| 4470 | 0.914 | 0.084 |
| … | … | … |
| 5689 | 0.923 | 0.078 |
Both SimLVSeg-3D and SimLVSeg-SI outperform the state-of-the-art,suggesting that the superior performance can be attributed to the SimLVSeg design rather than the selection of the underlying network architectures.
原作者在论文中说是SimLVSeg设计优秀所以性能较好而不是主干网络的选择,从移植SegMamba的实验结构来看确实如此。
改小参数量
具体的措施有:将通道数改为原来的1/2,并转为32位精度计算
加入通道注意力模块
序言
在本文中,作者研究了网络设计的一个不同方面-通道之间的关系。引入了一个新的架构单元,称之为挤压和激发(SE)块,其目标是通过显式建模其卷积特征通道之间的相互依赖性来提高网络产生的表示的质量。为此,作者提出了一种允许网络执行特征重新校准的机制,通过该机制,网络可以学习使用全局信息来选择性地强调信息特征并抑制不太有用的特征
在传统的CNN架构中,通常采用卷积层和池化层来提取图像特征。然而,这种方法并没有明确地对特征通道之间的关系进行建模,导致一些通道对于特定任务的贡献相对较小,而其他通道则更重要。SE模块旨在解决这个问题。
方法
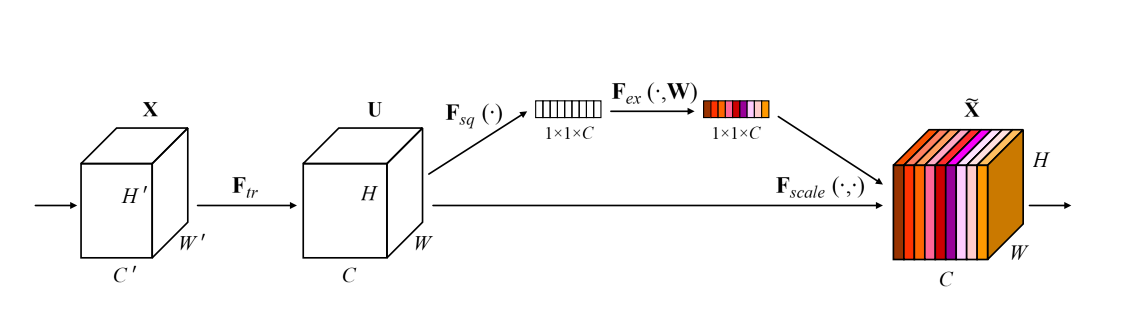
SE构建块的结构如图1所示。
Squeeze操作
这个操作对应上图中的Fsq函数。上面提到了,要找到通道之间的关系,那就是要用到所有通道的信息。论文中简单起见,把每个通道都压缩成一个点,直接求平均,相当于就是这个通道的特征值。这个求平均的过程就是所谓的Squeeze。表达为公式就是:
uc表示特征图U中的某个通道,zc表示经过压缩之后的向量Z。从公式中可以看出,每个尺寸为H*W的featrue map通道,经过全局平均之后,就只剩一个像素点。所以,一个的featrue map经过squeeze之后,就变成了一个的vector, 计作Z。
Excitation
特征图经过压缩后,第二步就是综合这些通道的特征值。论文提出,这个综合方法需要满足两个条件:1.要可以表达非线性关系 2.要可以提取处非互斥的关系 所以,论文提出了一个计算方法:
从上面可以看出,这个Excitation就是两个全联接层,第一个连接层把1* 1* C的vector Z变成一个1* 1* C/r的向量。然后后面跟一个ReLU激活 层。· 第二个连接层把1* 1* C/r的vector重新恢复为一个1* 1* C的向量。然后后面跟一个Sigmod 的激活层。
Scale
最后,根据图1,作为Squeeze和Excitation操作之后,还需要和原特征图U进行合并,也就是上面提到的“重新校准特征图”。这个合并方法也特别简单,就是把这个向量直接原特征图相乘。
从上面的excitation可以得知,s是一个的向量,与一个1* 1* C的特征图相乘的话,再从上面的公式可以看出,就是按通道数一个一个的去把s中的每个元素拿出来与特征图的对 应通道进行相乘,就是一个标量乘以矩阵,所以可以理解就是一个scale的操作,放大缩小而已。讲到这里,感觉就是SE就是提取出了通道中某些特征,或者说某些注意力特征,通过这个s’也就是表示通道的权重的向量,去把这个权重乘以对应的特征图通道,来放大或缩小这个通道的作用。从这个角度上来看,确实就叫做通道注意力机制。
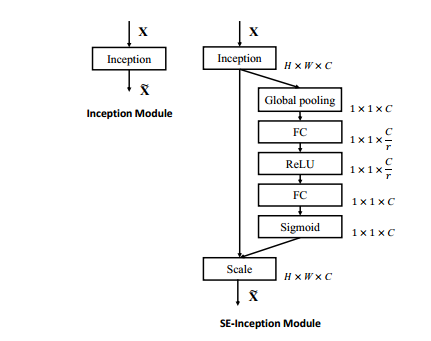
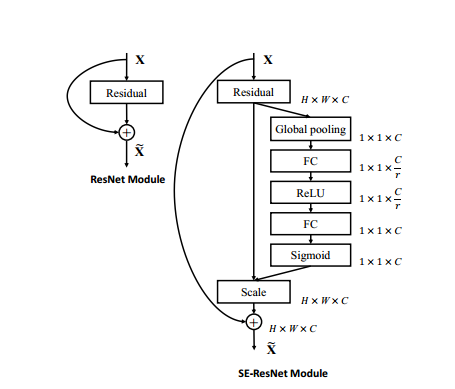
下面给出了SE与Inception和ResNet合并的方式。
总结
总而言之,SE模块通过引入Squeeze和Excitation操作,通过自适应地学习每个通道的权重,增强了神经网络的表达能力和性能,使网络能够更有效地学习和利用特征通道之间的关系。
通道再次降低为初始的1/3,即24M的模型,同时加入通道注意力模块,即上面的压缩激励
加入MSAA模块
加入多尺度注意力聚合模块MSAA
提出CM-UNet框架:基于Mamba架构的CM-UNet框架,通过整合CNN和Mamba模块,能够在遥感图像语义分割任务中高效捕捉局部和全局信息。
设计CSMamba块:CSMamba块结合了通道和空间注意力机制,将Mamba模块扩展为能够处理图像长程依赖的组件,提升了特征选择和信息融合的精度。
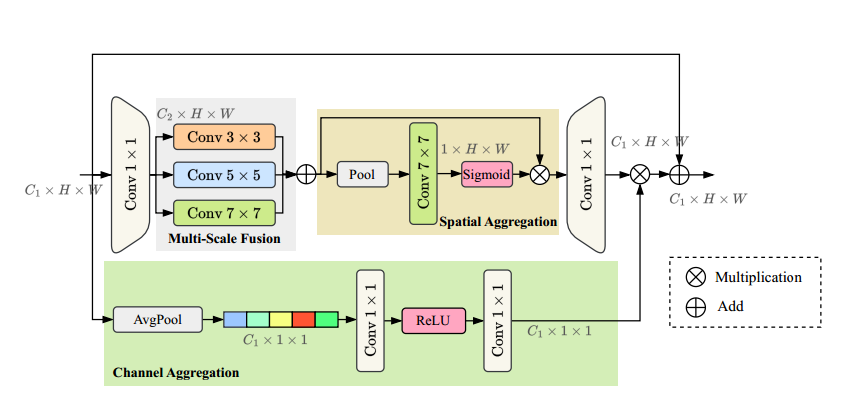
多尺度注意力聚合模块(MSAA):引入MSAA模块,聚合编码器的多尺度特征,通过空间和通道的双重聚合提高特征表达能力,替代传统的跳跃连接,更好地支持解码器的多层次信息融合。
多输出监督机制:在解码器的不同层次引入多输出监督,确保各层次逐步细化分割图,从而提升最终分割精度。
整体结构
CM-UNet模型结构由ResNet编码器、多尺度注意力聚合模块(MSAA)和CSMamba解码器组成。编码器负责提取多层次特征,MSAA模块融合多尺度特征以增强表达,解码器则利用CSMamba块通过通道和空间注意力机制高效捕捉长程依赖关系,最终生成精细的分割图,并在各层解码器中加入多输出监督以优化分割结果。
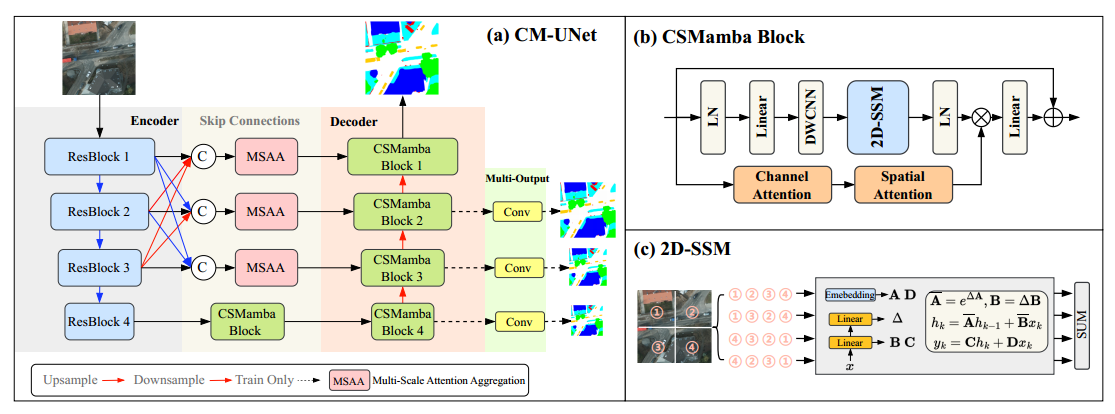
CNN编码器:采用ResNet结构作为编码器,用于提取多层次的特征信息。与传统UNet不同,CM-UNet的编码器使用的是多尺度特征提取,以便为后续模块提供更丰富的上下文信息。
多尺度注意力聚合模块(MSAA):在编码器和解码器之间,使用MSAA模块对多尺度特征进行聚合。这个模块通过空间和通道注意力机制对不同尺度的特征进行融合,增强特征表达能力,并取代了UNet中的跳跃连接。
CSMamba解码器:解码器采用CSMamba块,该模块结合通道和空间注意力机制,以及Mamba结构的线性时间复杂度特性,能够有效捕获图像的长程依赖关系。CSMamba解码器逐步上采样特征并生成输出分割图。同时引入多输出监督,在各层解码器中加入监督信号,确保分割图在不同层次上得到精细化生成。
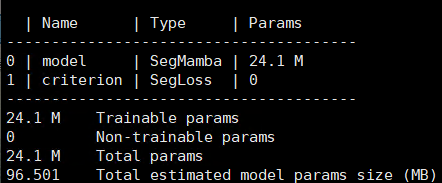
参数量降为24.1M
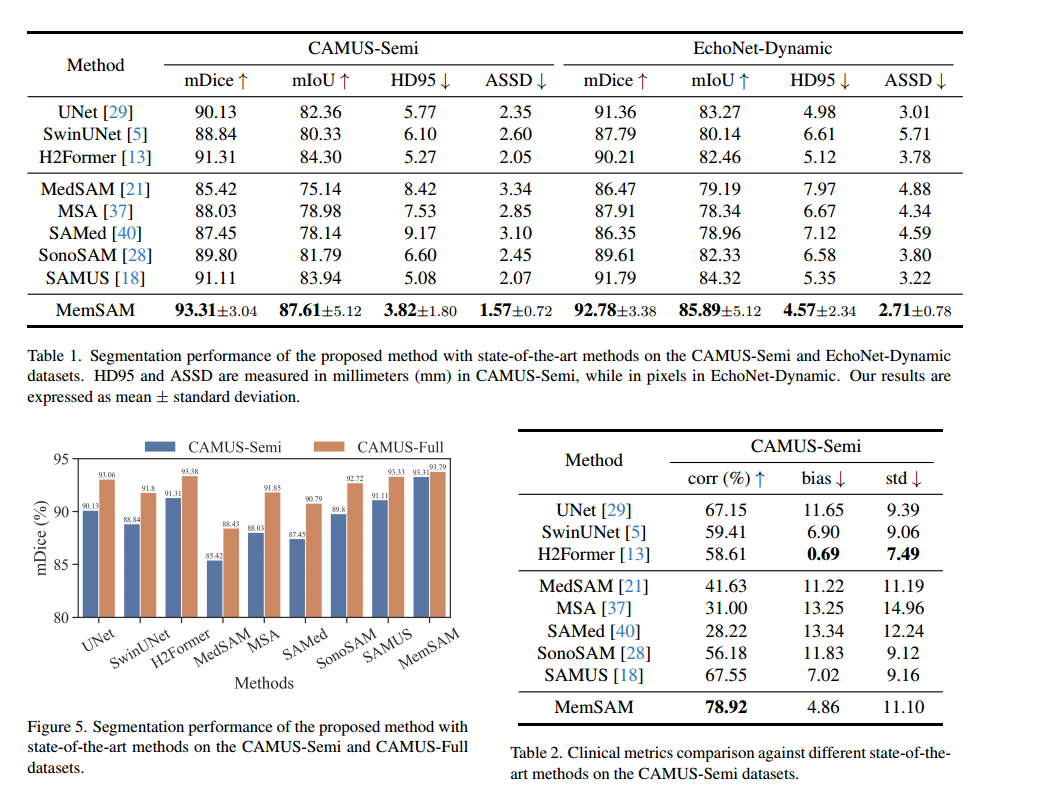
CVPR 2024 的MemSAM
Deng X, Wu H, Zeng R, et al. MemSAM: Taming Segment Anything Model for Echocardiography Video Segmentation[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. 2024: 9622-9631.
该论文实验结果
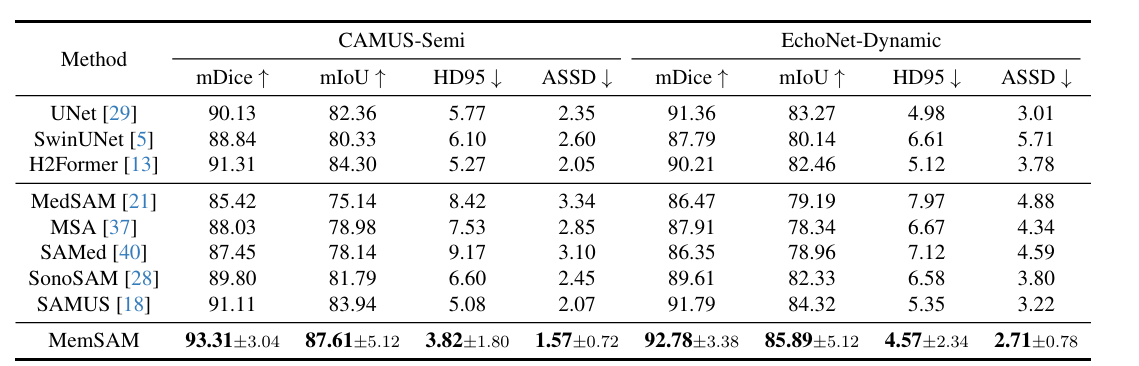
camus-semi和camus-full
后续实验参见后续数据集实验部分
2.2 模型修改-LightM-UNet
运行环境版本:
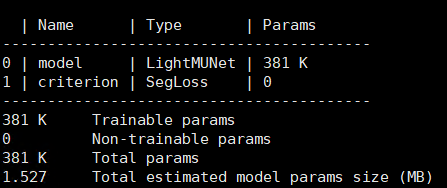
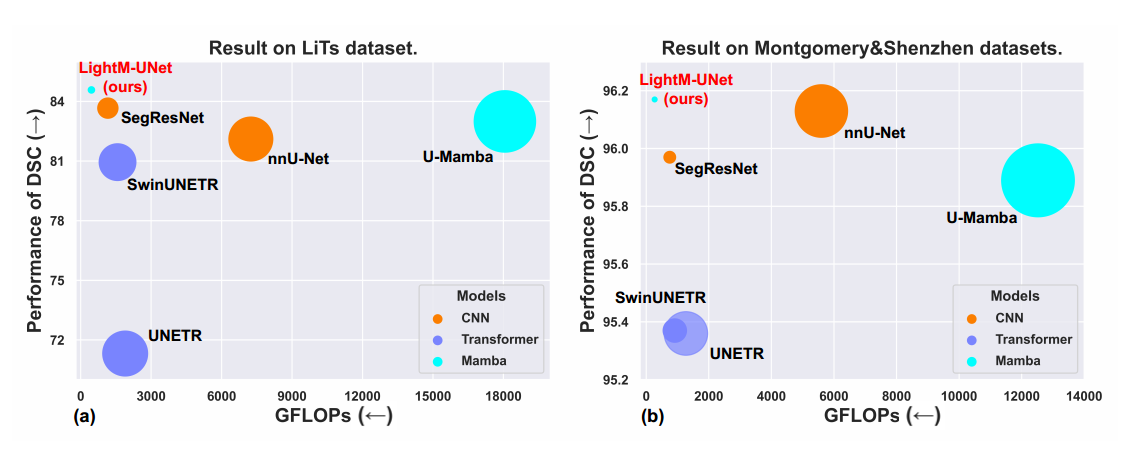
LightM-UNet,这是一个基于Mamba的轻量级U形分割模型,它在显著降低参数和计算成本的同时实现了最先进的性能,参数大大减小
所提出的LightM-UNet的总体架构如图所示。给定一个输入图像 ,其中 、、 和 分别表示3D医疗图像的通道数、高度、宽度和切片数
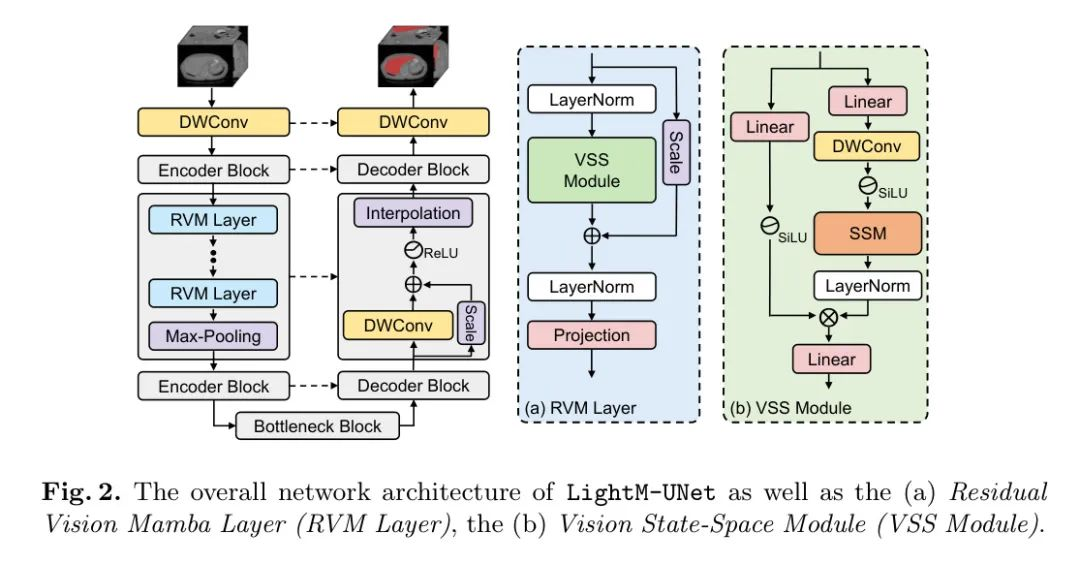
LightM-UNet首先使用深度可分卷积(DWConv)层进行浅层特征提取,生成浅层特征图。随后,LightM-UNet结合三个连续的编码器块(Encoder Blocks)从图像中提取深层特征。在每个编码器块之后,特征图中的通道数翻倍,而分辨率减半。
Encoder Block
感觉encoder都差不多
为了最小化参数数量和计算成本,LightM-UNet采用了仅包含Mamba结构的编码器块来从图像中提取深层特征。
具体来说,给定一个特征图 ,其中,,,,以及,编码器块首先将特征图展平并转置成的形状,其中。
随后,编码器块使用个连续的RVM层来捕捉全局信息,在最后一个RVM层中通道数增加。此后,编码器块重新调整并转置特征图的形状为 ,紧接着进行最大池化操作以降低特征图的分辨率。
最终,第l个编码器块输出新的特征图,其形状为。
Residual Vision Mamba Layer (RVM Layer)
LightM-UNet提出了RVM层以增强原始的SSM块,用于图像深层语义特征提取。具体来说,LightM-UNet利用先进的残差连接和调整因子进一步增强了SSM的长距离空间建模能力,几乎不引入新的参数和计算复杂性。
实验
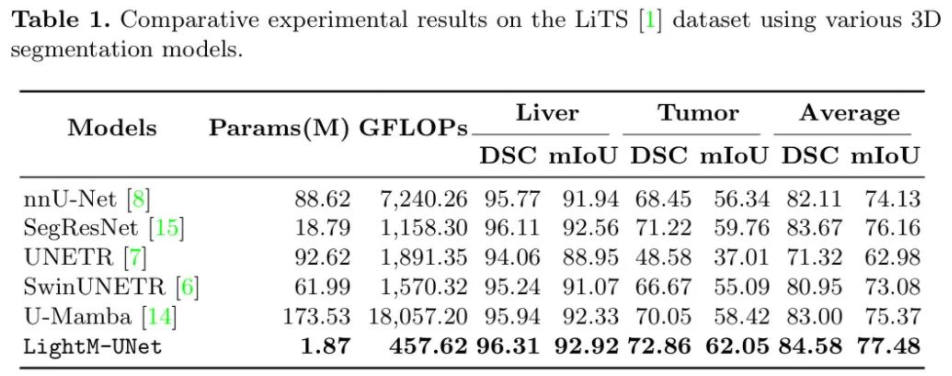
LightM-UNet以其极低的参数数量脱颖而出,仅使用了1.09M个参数。与nnU-Net和U-Mamba相比,参数分别减少了99.14%和99.55%
加入通道注意力特征融合
同时实验时加入mIOU评价指标
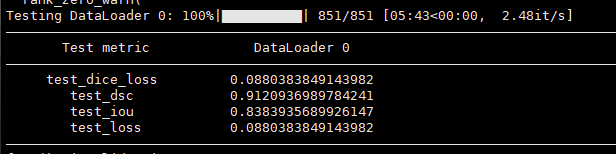
2.3 更改扫描方式
选择一:Right Diagonal (RD 扫描)

选择二:Right Diagonal Two-way(RD 双向扫描)
聚焦于图的中心
选择三:使用Hilbert曲线 (Hilbert 扫描)
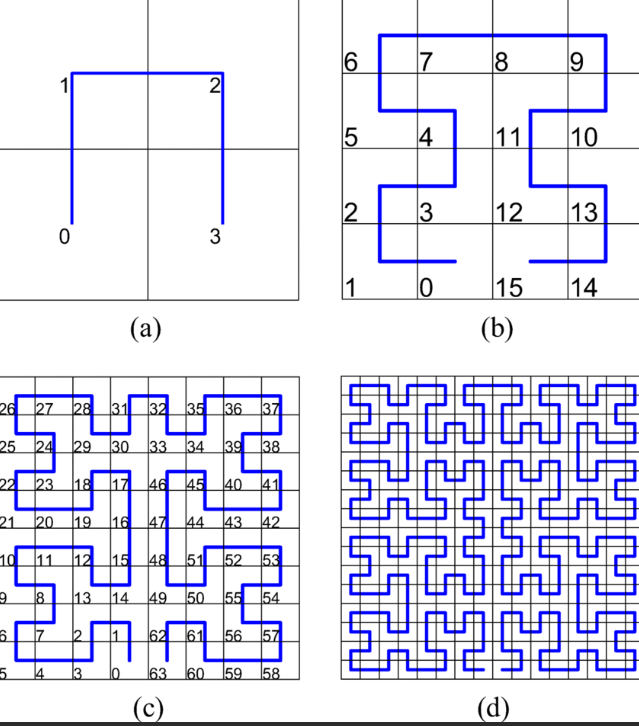
通过递归的分形结构,将高维空间(如二维平面)连续地映射到一维直线上,同时尽可能保持局部区域的邻近性,Hilbert曲线以及其离散近似表示方法都非常实用,因为其将多维空间转换为一维空间的方法很好地保留了空间邻近性。(x,y)是一个单元方格中的点,d代表该点在Hilbert曲线上的位置,而由于其空间的邻近性,在单元格上近似的点,其对应Hilbert的d值也比较接近。
要想更改扫描方式,因为该曲线只支持2的幂的边长,该数据为112 * 112 的规格,最为接近的2的幂为128。因此必须先得把数据集的数据填充到128 *128的规格,尝试了两种填充方式:
- 直接resize
- 使用0填充图像的右边和底边
其中直接resize可能导致一定程度上标签的错位,猜测会不如第二种使用0填充的方式。具体的实验数据可参看3.3 Echo-Pediatric实验部分。
跑一轮需要三百多个小时,继续优化
选择四:moore 曲线扫描
和希尔伯特曲线类似,是希尔伯特曲线的循环版本,不过是从中间开始,首尾闭合
3. 数据集实验
SAM的
3.1 EchoNet-Dynamic
小segmamba实验结果(DiceLoss)
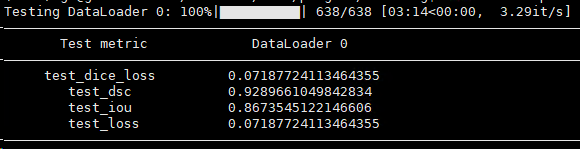
DICE和IOU都比该文高,其中DICE高0.11个点,IOU高0.84
其他的损失函数
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| diceloss | 0.9289 | 0.8673 | 0.0718 |
| jaccard | 0.9276 | 0.8650 | 0.1353 |
| jaccard | 0.9267 | 0.8635 | 0.1367 |
| tversky | 0.9272 | 0.8643 | 0.0687 |
效果一般
改为使用moore扫描
方向实验
| segmamba-MRscan | dsc | iou | loss |
|---|---|---|---|
| dice + Adaw_4bt-4lr 32F 0 + - v191 | 0.9323 | 0.8733 | |
| - v194 | 0.932656 | 0.873810 | |
| dice + Adaw_4bt-4lr 32F 3 + - v192 | 0.9321 | 0.8729 | |
| - v199 | 0.9325 | 0.8736 | |
| dice + Adaw_4bt-4lr 32F 2 + - v193 | 0.9325 | 0.8736 | |
| - v200 | 0.9323 | 0.8732 |
还是 0 + - 最好
学习率实验
| segmamba-MRscan | dsc | iou | loss |
|---|---|---|---|
| dice + Adaw_16bt-4lr 32F 0 + - | |||
| - | |||
| dice + Adaw_16bt-8lr 32F 0 + - | |||
| - | |||
| dice + Adaw_16bt-16lr 32F 0 + - | |||
| - |
损失实验
| segmamba-MRscan 0 + - | dsc | iou | loss |
|---|---|---|---|
| 0.8 dice + 0.2 jaccard + Adaw_4bt-4lr 32F v209 | 0.9321 | 0.8729 | 0.0798 |
| - v212 | 0.9327 | 0.8739 | 0.0792 |
| 0.4 dice + 0.3 jaccard + 0.3 focal + Adaw_4bt-4lr 32F v210 | 0.932578 | 0.873673 | 0.07737 |
| - v213 | 0.932525 | 0.873580 | 0.07741 |
| 0.2 dice + 0.2 jaccard + 0.6 focal + Adaw_4bt-4lr 32F v211 | 0.932560 | 0.873642 | 0.0561 |
| - v214 | 0.9324 | 0.8734 | 0.0562 |
| mean 0.3 dice + 0.1 jaccard + 0.3 tversky + 0.3 focal v230 | 0.9267 | 0.8658 |
3.2 CAMUS
重写dataset改为该数据集后,DICE和IOU都出现错误:表现为大于1。
猜想:每个病人只有十几帧,但是segmamba是按128帧设计的,因此输入的是128帧,多出的帧采用零填充,导致有用数据太少。
方案:使用每个病人原始的数据,进行重复,直到填满128帧。
并不是这个原因,是因为拼接的函数有点问题,改完之后可以正常显示数据,但是还不完善,只能使用一个batchsize
下面再来测试几种填充之后的实验差别
| 填充方式 | dsc | iou | loss |
|---|---|---|---|
| type1 | 0.9123 | 0.8459 | 0.0950 |
| type2 | 0.9301 | 0.8708 | 0.0779 |
| type3 | 0.9234 | 0.8603 | 0.0846 |
| type4 | 0.9265 | 0.8650 | 0.0859 |
综合来看type2的padding效果最好,后续在type2上继续实验
将所有的消融实验做一个表格,包括
| segmamba-24.1M | DICE | IOU | loss |
|---|---|---|---|
| type1 + Adaw + diceloss | 0.912 | 0.845 | 0.095 |
| type2 + Adaw + diceloss | 0.930 | 0.870 | 0.077 |
| type3 + Adaw + diceloss | 0.923 | 0.860 | 0.084 |
| type4 + Adaw + diceloss | 0.926 | 0.865 | 0.085 |
| type5 + Adaw + diceloss | 0.912 | 0.845 | 0.095 |
| type2 + Adaw + jaccard | 0.932 | 0.873 | 0.129 |
| type2 + Adaw + jaccard+ 2lr | 0.935 | 0.878 | 0.126 |
| type3 + Adaw + hccdice | 0.931 | 0.871 | 0.080 |
| type3 + Adaw + jaccard | 0.932 | 0.873 | 0.135 |
| type3 + Adaw + jaccard + 2lr | 0.934 | 0.877 | 0.125 |
| type3 + Adaw + jaccard + 3lr | 0.932 | 0.873 | 0.128 |
| type3 + Adaw + tversky + 2lr | 0.930 | 0.870 | 0.076 |
| type3 + SGD + diceloss | 0.921 | 0.857 | 0.078 |
| type3 + SGD + jaccard | 0.919 | 0.850 | 0.149 |
| type4 + Adaw + jaccard | 0.933 | 0.875 | 0.127 |
| type6 + Adaw + jaccard + 2lr | 0.928 | 0.866 | 0.140 |
使用CAMUS数据集官方划分的训练集,验证集和测试集
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| 全监督 | 0.9377 | 0.8828 | 0.1208 |
| 全监督_4bt-4lr | 0.9406 | 0.8879 | 0.1193 |
| 弱监督A | 0.9391 | 0.8852 | 0.1150 |
| 弱监督B | 0.2071 | 0.1155 | 0.9697 |
这里的弱监督A是训练的时候只提取了两帧(舒张末期和收缩末期),在验证测试时也只测试了这两帧;弱监督B也是训练的时候只提取两帧,但是验证和测试时是全周期数据,可以看到,效果非常的垃圾。。。
使用SAM模型划分的训练集,验证集和测试集
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| dice+Adaw_4bt-3lr | 0.9427 | 0.8917 | 0.0681 |
| jaccard+Adaw_4bt-4lr | 0.9431 | 0.8924 | 0.1143 |
| jaccard+Adaw_4bt-3lr | 0.9414 | 0.8893 | 0.1215 |
在3D_UNet上
| 3D_UNet | dsc | iou | loss |
|---|---|---|---|
| dice+Adaw_2lr+split_SAM | 0.9430 | 0.8922 | 0.0933 |
| dice+Adaw_4lr+split_SAM | 0.9457 | 0.8970 | 0.0633 |
| dice+Adaw_2lr+split_CAMUS | 0.9351 | 0.8782 | 0.0954 |
| dice+Adaw_4lr+split_CAMUS | 0.9440 | 0.8940 | 0.0631 |
改进hibertScan后 使用SAM的划分方式 segmamba-HBscan
| segmamba-HBscan | dsc | iou | loss |
|---|---|---|---|
| hccdice + SGD | 0.9346 | 0.8773 | 0.0655 |
| hccdice + Adaw_4bt-4lr | 0.9397 | 0.8863 | 0.0690 |
| jaccard + Adaw_4bt-4lr | 0.9376 | 0.8825 | 0.1283 |
| hccdice + Adaw_4bt-5lr | 0.9421 | 0.8906 | 0.0634 |
| hccdice + Adaw_4bt-6lr | 0.9402 | 0.8872 | 0.0651 |
| hccdice + Adaw_4bt-5.5lr | 0.9394 | 0.8858 | 0.0659 |
| tversky + Adaw_4bt-5lr | 0.9385 | 0.8842 | 0.0675 |
| tversky + Adaw_4bt-5lr type6 all 32F | 0.9430 | 0.8923 | 0.0580 |
| tversky + Adaw_4bt-5lr type2 all 32F | 0.9418 | 0.8900 | 0.0642 |
| hccdice + Adaw_4bt-5lr type6 all 32F | 0.9398 | 0.8865 | 0.0637 |
| hccdice + Adaw_8bt-7.07lr type6 32F | 0.9386 | 0.8844 | 0.0673 |
| hccdice + Adaw_12bt-8.65lr type6 32F | 0.9361 | 0.8800 | 0.0706 |
| hccdice + Adaw_4bt-5lr type2 EDES 32F | 0.9408 | 0.8882 | 0.0648 |
| hccdice + Adaw_4bt-5lr type2 all 32F | 0.9424 | 0.8911 | 0.0618 |
| hccdice + Adaw_16bt-5lr type2 all 32F | 0.9368 | 0.8812 | 0.0996 |
| hccdice + Adaw_16bt-15lr type2 all 32F | 0.9397 | 0.8863 | 0.0659 |
| hccdice + Adaw_16bt-20lr type2 all 32F | 0.9410 | 0.8886 | 0.0624 |
| hccdice + Adaw_16bt-25lr type2 all 32F | 0.9390 | 0.8850 | 0.0633 |
segmamba-MRscan
| segmamba-MRscan | dsc | iou | loss |
|---|---|---|---|
| tversky + Adaw_4bt-5lr type2 all 32F | 0.9420 | 0.8903 | 0.0612 |
| tversky + Adaw_4bt-5lr type6 all 32F | 0.9421 | 0,8906 | 0.0613 |
| hccdice + Adaw_4bt-5lr type2 all 32F | 0.9412 | 0.8890 | 0.0631 |
| hccdice + Adaw_4bt-5lr type6 all 32F | 0.9374 | 08823 | 0.0660 |
| jaccard + Adaw_4bt-5lr type2 all 32F | 0.9402 | 0.8872 | 0.1222 |
| jaccard + Adaw_4bt-5lr type6 all 32F | 0.9399 | 0.8866 | 0.1174 |
| dice + Adaw_4bt-5lr type2 all 32F | 0.9408 | 0.8883 | 0.0630 |
| dice + Adaw_4bt-5lr type6 all 32F | 0.9395 | 0.8859 | 0.0640 |
| tversky + SGD-0.1lr type2 all 32F | |||
| tversky + SGD-0.1lr type6 all 32F | |||
| tversky + Adaw_4bt-5lr type2 all 32F 35epoch | 0.9403 | 0.8874 | 0.0634 |
| tversky + Adaw_4bt-5lr type6 all 32F 35epoch | 0.9410 | 0.8885 | 0.0615 |
采用不同扫描方向实验这里都统一采用 tversky + Adaw_4bt-5lr type6 all 32F 60Epoch 0:西 1:北 2:东 3:南
第一轮的数据可能是没加MRscan的数据。。。
| segmamba-MRscan | dsc | iou | loss |
|---|---|---|---|
| 0 + - * | 0.9427 / 0.9408/0.9396/0.9413 | / | / |
| 1 + - | 0.9367 / 0.9401/0.9400/0.9394 | 0.8868 | 0.0635 |
| 2 + - * | 0.9416 / 0.9435/0.9421/0.9426 | 0.8931 | 0.0589 |
| 3 + - * | 0.9425 / 0.9418/0.9426/0.9421 | 0.8914 | 0.0602 |
| 0 - + * | 0.9418 / 0.9405/0.9421/0.9416 | 0.8906 | 0.0597 |
| 1 - + | 0.9385 / 0.9407/0.9405/0.9384 | / | / |
| 2 - + | 0.9401 / 0.9413/0.9391/0.9401 | 0.8892 | 0.0617 |
| 3 - + | 0.9410 / 0.9413/0.9391/0.9410 | / | / |
消融实验 减去MRscan 采用 tversky + Adaw_4bt-5lr type6 all 32F 60Epoch
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| tversky + Adaw_4bt-5lr type2 all 32F | 0.9438 | / | / |
| tversky + Adaw_4bt-5lr type6 all 32F | 0.9434 | 0.8929 | 0.059 |
| tversky + Adaw_4bt-5lr type2 all 32F 35epoch | |||
| tversky + Adaw_4bt-5lr type6 all 32F 35epoch | 0.9409 | 0.8884 | 0.0636 |
| 后面统一采用tversky + Adaw_4bt-5lr type6 all 32F 60Epoch | |||
| tversky + Adaw_4bt-5lr type6 all 32F 60Epoch | 0.9407 | 0.8881 | 0.0621 |
| tversky + Adaw_4bt-5lr type6 all 32F 60Epoch | 0.9409 | 0.8884 | 0.0614 |
| tversky + Adaw_4bt-5lr type6 all 32F 60Epoch | 0.9420 | 0.8904 | 0.0617 |
| tversky + Adaw_4bt-5lr type6 all 32F 60Epoch |
学习率实验
| segmamba-MRscan 2 + - | dsc | iou | loss |
|---|---|---|---|
| tversky + Adaw_4bt-4lr type6 all 32F | 0.942372 | 0.8910 | 0.0631 |
| - | 0.941797 | 0.8899 | 0.0627 |
| tversky + Adaw_4bt-5lr type6 all 32F | 0.941884 | 0.8901 | 0.0606 |
| - | 0.941728 | 0.8898 | 0.0621 |
损失实验 mean
| segmamba-MRscan 2 + - | dsc | iou | loss |
|---|---|---|---|
| tversky | 0.9391 | 0.8864 | |
| - | 0.9392 | ||
| 0.3 dice + 0.1 jaccard + 0.3 tversky + 0.3 focal v228 | 0.9391 | 0.8863 | |
| - v229 | 0.9339 | 0.8772 |
CAMUS_filled_32_trunc_8 实验
| dsc | iou | ASSD | HD95 | |
|---|---|---|---|---|
| v457 | 0.9419 | 0.8903 | ||
| v461 | 0.8867 | 0.7965 | ||
| v462 | 0.8902 | 0.8022 |
分相位的指标
| Model | Phase | DSC | IOU | ASSD | HD95 |
|---|---|---|---|---|---|
| 3D U‑Net | ED | 91.97 | 85.29 | 1.47 | 3.96 |
| ES | 87.87 | 79.03 | 1.86 | 5.17 | |
| SegFormer3D | ED | 92.90 | 86.86 | 1.27 | 3.32 |
| ES | 89.14 | 80.91 | 1.57 | 4.07 | |
| UKAN3D | ED | 91.50 | 84.80 | 1.54 | 3.96 |
| ES | 88.29 | 79.46 | 1.68 | 3.82 | |
| EM‑Mamba | ED | 93.31 | 87.55 | 1.20 | 2.88 |
| ES | 90.87 | 83.62 | 1.30 | 3.25 |
3.3 Echo-Pediatric
该数据需要首先进行预处理,使用split列中的数字划分训练集,验证集和测试集。这里使用的是<8的数据为训练集,==8的为验证集,其余的为测试集。
除此之外,该数据集中有部分脏数据需要进行处理,比如VolumeTracings.csv中部分X,Y数据为空,Frame为No Systolic,需要删除,以及部分数据标注时有误,只有144帧,但标注时标注了序号为第144帧,导致越界错误。
先使用了jaccard的dsc只有0.9063,iou为0.8288;考虑到这个数据集和echodynamic类似。因此,接着使用diceloss进行测试,效果差不多,dsc为0.9045,iou为0.8256
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| jaccard+Adaw | 0.9063 | 0.8288 | 0.0938 |
| diceloss+Adaw | 0.9045 | 0.8256 | 0.1748 |
数据集的处理有点问题,重新跑一遍:
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| jaccard+Adaw_4lr | 0.9039 | 0.8246 | 0.1757 |
| jaccard+Adaw_6lr | 0.9041 | 0.8250 | 0.1752 |
| jaccard+SGD | 0.9084 | 0.8323 | 0.1677 |
| diceloss+SGD | 0.9068 | 0.8295 | 0.0932 |
| hccdice+Adaw_4lr | 0.9115 | 0.8374 | 0.0892 |
3D_Unet模型 Adaw
| 3D_Unet | dsc | iou | loss |
|---|---|---|---|
| 3D_Unet+jaccard | 0.9090 | 0.8332 | 0.1688 |
| 3D_Unet+diceloss | 0.9112 | 0.8369 | 0.0906 |
其他模型 都是Adaw+DiceLoss
| model name | dsc | iou | loss |
|---|---|---|---|
| LightMUNet | 0.8855 | 0.7946 | 0.1145 |
| TMamba3D | / | / | / |
| SegFormer3D | 0.9123 | 0.8388 | 0.0876 |
| UKAN3D | 0.9091 | 0.8334 | 0.0910 |
UNet_IDC3D模型
| model name | dsc | iou | loss |
|---|---|---|---|
| UNet_IDC3D+diceLoss | 0.9112 | 0.8368 | / |
| UNet_IDC3D+hccdice | 0.9137 | 0.8412 | 0.0866 |
改进为希尔伯特曲线扫描后的segmamba-HBscan
填充到128*128规格的两种方式的实验
| segmamba-24.1M | dsc | iou | loss |
|---|---|---|---|
| hccdice+Adaw_4lr (resize 128) | 0.9084 | 0.8323 | 0.0923 |
| hccdice+Adaw_4lr (padding 128) | 0.9122 | 0.8386 | 0.0884 |
| hccdice + Adaw_4bt-5lr 32F | 0.9137 | 0.8412 | 0.0870 |
| tversky + Adaw_4bt-5lr 32F | 0.9105 | 0.8357 | 0.0835 |
segmamba-HBscan
| segmamba-HBscan | dsc | iou | loss |
|---|---|---|---|
| hccdice+Adaw_4lr | 0.9126 | 0.8393 | 0.0882 |
| jaccard+Adaw_4lr | 0.9115 | 0.8374 | 0.1628 |
| hccdice+Adaw_4bt-5lr 32F | 0.9139 | 0.8415 | 0.0868 |
| hccdice+SGD_4bt-0.1lr | 0.9128 | 0.8396 | 0.0877 |
| hccdice+SGD_16bt-0.3lr 32F | / | / | / |
| tversky + Adaw_4bt-5lr 32F | / | / | / |
moore曲线的扫描方式 segmamba-MRScan
| segmamba-MRscan | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-5lr 32F | 0.9110 | 0.8367 | 0.0898 |
| tversky + Adaw_4bt-5lr 32F | 0.9104 | 0.8356 | 0.0809 |
更换了不同的扫描方向
| segmamba-MRscan 2 + - | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-5lr 32F | 0.9112 | 0.8370 | 0.0897 |
| - | 0.9119 | 0.8381 | 0.8882 |
| - | 0.9113 | 0.8371 | 0.0895 |
| hccdice + Adaw_4bt-4lr 32F | 0.913840 | 0.8413 | 0.0870 |
| - | 0.9122 | 0.8386 | 0.0886 |
| hccdice + Adaw_4bt-6lr 32F | 0.9124 | 0.8389 | 0.0884 |
| hccdice + SGD_ 4bt-0.1lr 32F | 0.913801 | 0.8412 | 0.0866 |
| hccdice + SGD_ 4bt-0.15lr 32F | 0.9085 | 0.8324 | 0.09198 |
| segmamba-MRscan 3 + - | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-4lr 32F | 0.9135 | 0.8409 | 0.0871 |
| - | 0.9142 | 0.8420 | 0.0866 |
| hccdice + SGD_ 4bt-0.1lr 32F | 0.9120 | 0.8383 | 0.0884 |
| - | 0.9044 | 0.8255 | 0.0964 |
| segmamba-MRscan 0 + - | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-4lr 32F | 0.9148 | 0.8430 | 0.0859 |
| - | 0.9149 | 0.8431 | 0.0859 |
| hccdice + SGD_ 4bt-0.1lr 32F | 0.9082 | 0.8319 | 0.0923 |
| - | 0.9127 | 0.8395 | 0.0876 |
| segmamba-MRscan 0 - + | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-4lr 32F | 0.9126 | 0.8393 | 0.0884 |
| - | 0.9115 | 0.8375 | 0.0895 |
| hccdice + SGD_ 4bt-0.1lr 32F | 0.9131 | 0.8402 | 0.0872 |
| - | / | / | / |
| segmamba-MRscan 3 - + | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-4lr 32F | 0.9120 | 0.8383 | 0.0887 |
不同的损失测试
| segmamba-MRscan 0 + - | dsc | iou | loss |
|---|---|---|---|
| hccdice + Adaw_4bt-4lr 32F | 0.9114 | 0.8373 | 0.0895 |
| tversky + Adaw_4bt-4lr 32F | 0.9093 | 0.8337 | 0.0861 |
| - | 0.9089 | 0.8331 | 0.0848 |
| dice + Adaw_4bt-4lr 32F | 0.9151 | 0.8434 | 0.0852 |
| - | 0.9151 | 0.8435 | 0.0851 |
| jaccard + Adaw_4bt-4lr 32F v179 | 0.916061 | 0.8451 | 0.1551 |
| - | 0.9158 | 0.8448 | 0.1556 |
| focal + Adaw_4bt-4lr 32F | 0.9133 | 0.8405 | 0.0096 |
| - | 0.9153 | 0.8439 | 0.0091 |
| 0.5 dice+0.5 Jaccard + Adaw_4bt-4lr 32F | 0.9134 | 0.8407 | 0.1233 |
| - | 0.915999 | 0.8450 | 0.1198 |
| 0.8 dice+ 0.2 Jaccard + Adaw_4bt-4lr 32F | 0.9167 | 0.8462 | 0.0976 |
| - | 0.9159 | 0.8448 | 0.0986 |
| 0.2 dice+ 0.8 Jaccard + Adaw_4bt-4lr 32F | 0.9160 | 0.8451 | 0.1410 |
| - | 0.9137 | 0.8412 | 0.1446 |
| 0.4 dice + 0.3 jaccard + 0.3 focal + Adaw_4bt-4lr 32F v201 | 0.9165 | 0.8458 | 0.0974 |
| - v205 | 0.9157 | 0.8445 | 0.0985 |
| 0.6 dice + 0.2 jaccard + 0.2 focal + Adaw_4bt-4lr 32F v202 | 0.9145 | 0.8426 | 0.0961 |
| v206 | 0.9117 | 0.8378 | 0.0993 |
| 0.2 dice + 0.6 jaccard + 0.2 focal + Adaw_4bt-4lr 32F v203 | 0.9143 | 0.8422 | 0.1261 |
| - v207 | 0.9152 | 0.8436 | 0.1250 |
| 0.2 dice + 0.2 jaccard + 0.6 focal + Adaw_4bt-4lr 32F v204 | 0.9163 | 0.8455 | 0.0719 |
| - v208 | 0.9161 | 0.8452 | 0.0727 |
| (mean)0.3 dice + 0.1jaccard + 0.3 tversky + 0.3 focal v231 | 0.9079 | 0.8359 | |
| (mean)- v232 | 0.9071 | 0.8340 |
4. sota实验
4.1 Echo-Dynamic
| model name | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| 3d u-net v304 | 93.22 | 87.30 | 1.12 | 2.74 |
| SwinUnet | 87.79 | 80.14 | 5.71 | 6.61 |
| SegFormer3D v305 | 93.09 | 87.07 | 1.13 | 2.73 |
| H2Former | 90.21 | 82.46 | 5.12 | 3.78 |
| LightMUNet v348 | 92.67 | 86.34 | 1.21 | 2.95 |
| UKAN3D v341 | 93.20 | 87.27 | 1.12 | 2.67 |
| SegMamba | 92.88 | 86.71 | 1.73 | 5.30 |
| LKM-Unet | 91.18 | 83.79 | 2.84 | 9.91 |
| log-vmamba | 91.13 | 83.71 | 1.23 | 3.01 |
| MedSAM | 86.47 | 79.19 | 4.88 | 7.97 |
| SAMUS | 91.79 | 84.32 | 3.22 | 5.35 |
| MemSAM | 92.78 | 85.89 | 2.71 | 4.57 |
| ours | 93.28 | 87.41 | 1.10 | 2.68 |
| UKAN3D | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| v308 | 93.24 | 87.35 | / | / |
| v322 | 93.24 | 87.34 | 1.14 | 2.79 |
| v341 | 93.20 | 87.27 | 1.12 | 2.69 |
| v344 | 93.21 | 87.29 | 1.12 | 2.71 |
| v345 | 93.26 | 87.37 | 1.11 | 2.70 |
| LightMUnet | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| v306 | 92.80 | 86.57 | 1.18 | 2.91 |
| v348 | 92.67 | 86.34 | 1.21 | 2.95 |
| SegMamba | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V437 | 93.08 | 87.05 | ||
| V446 | 92.88 | 86.71 | 1.73 | 5.30 |
| LKM-Unet | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V442 | 91.18 | 83.79 | 2.84 | 9.91 |
| Log-vmamba | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V449 | 91.13 | 83.71 | 1.23 | 3.01 |
4.1 CAMUS
| model name | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| 3d u-net v321 | 93.78 | 88.28 | 2.82 | 8.93 |
| SwinUnet | 88.84 | 80.33 | 2.60 | 6.10 |
| SegFormer3D v312 | 93.46 | 87.72 | 2.13 | 4.27 |
| H2Former | 91.31 | 84.30 | 2.05 | 5.27 |
| LightMUNet v314 | 92.35 | 85.79 | 1.60 | 3.67 |
| UKAN3D v347 | 93.88 | 88.46 | 1.74 | 3.84 |
| SegMamba | 91.72 | 84.71 | 1.69 | 3.87 |
| LKM-Unet | 90.76 | 83.09 | 1.93 | 4.03 |
| Log-vmamba | 86.85 | 76.76 | 1.94 | 4.75 |
| MedSAM | 85.42 | 75.14 | 3.34 | 8.42 |
| SAMUS | 91.11 | 83.94 | 2.07 | 5.08 |
| MemSAM | 93.31 | 87.61 | 1.57 | 3.82 |
| ours | 94.30 | 89.21 | 1.36 | 2.93 |
| SegFormer3D | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| v312 | 93.46 | 87.72 | 2.13 | 4.27 |
| v463 | 92.96 | 86.86 |
| UKAN3D | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| v325 | 93.66 | 88.08 | 3.19 | 11.6 |
| v347 | 93.88 | 88.46 | 1.74 | 3.84 |
| SegMamba | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V432 | 93.34 | 87.51 | ||
| V433 | 91.72 | 84.71 | 1.69 | 3.87 |
| LKM-Unet | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V445 | 90.76 | 83.09 | 1.93 | 4.03 |
| Log-vmamba | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V452 | 86.85 | 76.76 | 1.94 | 4.75 |
4.3 Echo-Pediatric
| model name | dsc | iou | Param(M) | GFLOPS(G) | FPS |
|---|---|---|---|---|---|
| U-Net | 88.62 | 80.74 | 23.63 | / | / |
| SwinUnet | 89.64 | 81.05 | 62.19 | 99.05 | 35.90 |
| 3d u-net v41 | 90.90 | 83.32 | 18.83 | 29.44 | 96.29 |
| UKAN3D V48 | 90.92 | 83.35 | 9.12 | 28.59 | 93.58 |
| SegFormer3D v50 | 91.23 | 83.88 | 7.50 | 13.92 | 136.41 |
| H2Former | 88.53 | 80.41 | 33.71 | 32.23 | 140.38 |
| SegMamba | 90.92 | 83.35 | 67.40 | 195.98 | 28.39 |
| LKM-Unet | 91.49 | 84.32 | 14.22 | 1113.96 | 30.85 |
| Log-vmamba | 90.47 | 82.60 | 21.00 | 392.96 | 68.29 |
| ours | 92.43 | 85.93 | 24.10 | 23.73 | 42.09 |
| segmamba | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| V430 | 92.35 | 85.80 | ||
| V434 | 91.36 | 84.09 | ||
| V438 | 90.92 | 83.35 |
| LKM-Unet | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| V441 | 91.92 | 85.05 | ||
| V444 | 91.49 | 84.32 |
| Log-vmamba | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V450 | 90.47 | 82.60 |
| model name | dsc | iou | Param(M) | GFLOPS(G) | FPS |
|---|---|---|---|---|---|
| 3d u-net v41 | 0.9090 | 0.8332 | 18.83 | 29.44 | 96.29 |
| LightMUNet V45 | 0.8856 | 0.7946 | |||
| SegFormer3D v50 | 0.9123 | 0.8388 | |||
| UKAN3D V48 | 0.9092 | 0.8335 | |||
| ours | 0.9243 | 0.8593 | 24.10 | 23.73 | 42.09 |
v2
| model name | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| 3d u-net v331 | 91.34 | 84.06 | 1.64 | 4.02 |
| SegFormer3D v328 | 91.23 | 83.87 | 1.60 | 3.91 |
| LightMUNet v327 | 90.85 | 83.24 | 1.70 | 4.28 |
| UKAN3D v330 | 91.48 | 84.30 | 1.58 | 3.91 |
| ours | 91.65 | 84.58 | 1.55 | 3.85 |
| 3d u-net | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| v326 | 0.9156 | 0.8444 | / | / |
| v331 | 0.9155 | 0.8442 | 1.8086 | 4.9218 |
| v332 | 0.9158 | 0.8446 | / | / |
| v336 | 0.9134 | 0.8406 | 1.64 | 4.02 |
| UKAN3D | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| v329 | 0.9157 | 0.8445 | / | / |
| v330 | 0.9154 | 0.8444 | 1.6026 | 3.9676 |
| v337 | 0.9148 | 0.8430 | 1.5821 | 3.9149 |
4.4 显著性
echo-dynamic
| model name | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| 3d u-net v304 | 93.05 - 93.38 | 87.05 - 87.55 | 1.10 - 1.14 | 2.66 - 2.82 |
| SegFormer3D v305 | 92.94 - 93.24 | 86.83 - 87.31 | 1.11 - 1.15 | 2.68 - 2.78 |
| UKAN3D v341 | 93.04 - 93.36 | 87.02 - 87.52 | 1.10 - 1.14 | 2.61 - 2.73 |
| mooremamba | 93.13 - 93.43 | 87.17 - 87.65 | 1.09 - 1.13 | 2.62 - 2.74 |
camus
| model name | dsc | iou | assd | HD95 |
|---|---|---|---|---|
| 3d u-net v321 | 92.81 - 94.75 | 86.79 - 89.77 | 2.59 - 3.05 | 7.35 - 10.61 |
| SegFormer3D v312 | 92.09 - 94.82 | 85.91 - 89.53 | 1.17 - 3.09 | 3.645 - 4.90 |
| UKAN3D v347 | 93.26 - 94.50 | 87.44 - 89.48 | 1.69 - 1.79 | 3.65 - 4.03 |
| mooremamba V268 | 93.77 - 94.83 | 88.31 - 90.11 | 1.31 - 1.40 | 2.78 - 3.08 |
5. 消融实验
- 24.1 baseline
- moore扫描模块
- 融合损失
表格示意
+ fusionLoss: 0.4 dice + 0.3 jaccard + 0.3 focal
+ fusionLossV2: 0.4 dice + 0.1 jaccard + 0.5 focal (舍)
-fusionLoss: dice
+mooreScan: mooreScan
-mooreScan: 横向扫描resize
5.1 Echo-Dynamic
选用方向: 0 + -
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss (baseline) | 0.9213 | 0.8542 | 1.2950 | 3.5227 |
| + mooreScan - fusionLoss | 0.9324 | 0.8734 | 1.1086 | 2.6753 |
| - mooreScan + fusionLoss | 0.9317 | 0.8722 | 1.1223 | 2.7157 |
| + mooreScan + fusionLoss | 0.9328 | 0.8741 | 1.1050 | 2.6789 |
| - mooreScan - fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v251 | 0.9321 | 0.8728 | 1.1146 | 2.7019 | 0.9347 |
| v255 | 0.9320 | 0.8727 | 1.1144 | 2.7012 | 0.9345 |
| v286 | 0.9316 | 0.8720 | 1.1216 | 2.7196 | 0.9338 |
| v30 | 0.9276 | 0.8650 | |||
| v33 | 0.9272 | 0.8643 | |||
| v357 | 0.9259 | 0.8620 | 1.3182 | 3.5017 | |
| V366 | 0.9213 | 0.8542 | 1.2950 | 3.5227 |
| + mooreScan - fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v191 | 0.9323 | 0.8733 | 1.1086 | 2.6854 | 0.9337 |
| v249 | 0.9324 | 0.8734 | 1.1086 | 2.6753 | 0.9350 |
| - mooreScan + fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v248 | 0.9325 | 0.8737 | / | / | / |
| v252 | 0.9317 | 0.8722 | 1.1223 | 2.7157 | 0.9380 |
| v254 | 0.9325 | 0.8736 | 1.1086 | 2.6940 | 0.9356 |
b4 val 0.25
| + mooreScan + fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v210 | 0.9326 | 0.8737 | / | / | / |
| v246 | 0.9327 | 0.8739 | 1.1053 | 2.6756 | 0.9374 |
| v253 | 0.9328 | 0.8741 | 1.1050 | 2.6789 | 0.9368 |
| - mooreScan + fusionLossV2 | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v262 | 0.9326 | 0.8737 | / | / | / |
| v263 | 0.9322 | 0.8731 | / | / | / |
| + mooreScan + fusionLossV2 | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v256 | 0.9322 | 0.8730 | / | / | / |
| v257 | 0.9326 | 0.8737 | / | / | / |
5.2 CAMUS
选用方向:0 + -
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss (baseline) | 0.9266 | 0.8632 | 1.9351 | 3.8131 |
| + mooreScan - fusionLoss | 0.9416 | 0.8896 | 1.3347 | 2.8828 |
| - mooreScan + fusionLoss | 0.9393 | 0.8855 | 1.3134 | 2.9897 |
| + mooreScan + fusionLoss (ours) | 0.9430 | 0.8921 | 1.3594 | 2.9324 |
| - mooreScan - fusionLoss (baseline) | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v271 | 0.9416 | 0.8897 | / | / | / |
| v273 | 0.9412 | 0.8890 | / | / | / |
| V284 | 0.9376 | 0.8824 | 1.4344 | 3.1161 | 0.9690 |
| v285 | 0.9392 | 0.8853 | 1.4322 | 3.1237 | 0.9731 |
| v11 | 0.9123 | 0.8459 | |||
| v10 | 0.9301 | 0.8708 | |||
| v12 | 0.9234 | 0.8603 | |||
| v13 | 0.9265 | 0.8650 | |||
| v362 | 0.9212 | 0.8538 | |||
| v363 | 0.9280 | 0.8658 | 1.7142 | 3.5434 | |
| v364 | 0.9257 | 0.8617 | 1.7747 | 3.6552 | |
| v371 | 0.9345 | 0.8772 | |||
| v372 | 0.9318 | 0.8723 | |||
| v373 | 0.9266 | 0.8632 | 1.9351 | 3.8131 |
| + mooreScan - fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v274 | 0.9404 | 0.8875 | / | / | / |
| v275 | 0.9410 | 0.8885 | / | / | / |
| v276 | 0.9416 | 0.8896 | 1.3347 | 2.8828 | 0.9708 |
| v280 | 0.9415 | 0.8895 | 1.3756 | 3.1272 | 0.9717 |
| - mooreScan + fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v277 | 0.9428 | 0.8918 | / | / | / |
| v278 | 0.9393 | 0.8855 | 1.3134 | 2.9897 | 0.9644 |
| v281 | 0.9428 | 0.8918 | 1.3577 | 2.8974 | 0.9456 |
| v283 | 0.9402 | 0.8871 | 1.3599 | 2.9843 | 0.9688 |
| + mooreScan + fusionLoss (ours) | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v233 | 0.9394 | 0.8859 | 1.3565 | 2.9882 | 0.9674 |
| v234 | 0.9423 | 0.8909 | 1.3285 | 2.8963 | 0.9715 |
| v270 | 0.9419 | 0.8902 | / | / | / |
| v268 | 0.9430 | 0.8921 | 1.3594 | 2.9324 | 0.9732 |
| v269 | 0.9424 | 0.8912 | 1.3493 | 2.9682 | 0.9729 |
5.3 Echo-Pediatric
选用方向0 + -
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss (baseline) | 0.9137 | 0.8411 | 1.7467 | 4.4969 |
| + mooreScan - fusionLoss | 0.9218 | 0.8550 | 1.6444 | 3.9509 |
| - mooreScan + fusionLoss | 0.9231 | 0.8572 | 1.6132 | 3.9279 |
| + mooreScan + fusionLoss | 0.9243 | 0.8593 | 1.5865 | 3.9167 |
| - mooreScan - fusionLoss (baseline) | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v241 | / | / | / | / | / |
| v242 | 0.9145 | 0.8425 | |||
| v245 | 0.9137 | 0.8411 | 1.7467 | 4.4969 | 0.9267 |
| v38 | 0.9063 | 0.8288 | |||
| v39 | 0.9045 | 0.8256 | |||
| v42 | 0.9039 | 0.8246 | |||
| v43 | 0.9041 | 0.8250 | |||
| v352 | 0.9101 | 0.8351 | |||
| v353 | 0.9004 | 0.8188 | |||
| v354 | 0.9048 | 0.8262 | 1.7822 | 4.4320 | |
| v355 | 0.9052 | 0.8268 | 1.7902 | 4.4417 | |
| v367 | 0.9229 | 0.8568 |
| + mooreScan - fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v235 | 0.9138 | 0.8413 | 1.6069 | 3.9439 | 0.9303 |
| v236 | 0.9151 | 0.8436 | 1.5794 | 3.9623 | 0.9247 |
| v374 | 0.9218 | 0.8550 | 1.6444 | 3.9509 |
| - mooreScan + fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v239 | 0.9150 | 0.8433 | 1.5740 | 3.8816 | 0.9266 |
| v240 | 0.9151 | 0.8435 | / | / | / |
| v243 | 0.9148 | 0.8430 | 1.5646 | 3.8466 | 0.9229 |
| v356 | 0.9112 | 0.8368 | 1.6730 | 4.2085 | |
| v375 | 0.9241 | 0.8589 | 1.5877 | 3.9464 | |
| v376 | 0.9178 | 0.8481 | 1.6531 | 4.0372 | |
| v377 | 0.9231 | 0.8572 | 1.6132 | 3.9279 |
| + mooreScan + fusionLoss | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v216 | 0.9084 | 0.8367 | / | / | / |
| v217 | 0.9073 | 0.8352 | / | / | / |
| v201 | 0.9165 | 0.8458 | 1.5507 | 3.8580 | 0.9250 |
| v266 | 0.9141 | 0.8418 | / | / | / |
| v267 | 0.9137 | 0.8411 | / | / | / |
| v368 | 0.9243 | 0.8593 | 1.5865 | 3.9167 |
| - mooreScan + fusionLossV2 | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v260 | 0.9160 | 0.8450 | 1.5611 | 3.8918 | 0.9167 |
| v261 | 0.9163 | 0.8455 | 1.5586 | 3.8783 | 0.9237 |
| V264 | 0.9154 | 0.8440 | / | / | / |
| V265 | 0.9162 | 0.8454 | / | / | / |
| + mooreScan + fusionLossV2 | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v258 | 0.9161 | 0.8451 | / | / | / |
| v259 | 0.9148 | 0.8430 | 1.5939 | 3.9800 | 0.9208 |
5.4 显著性
echo-dynamic
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss | 91.98 - 92.28 | 85.18 - 85.66 | 1.28 - 1.31 | 3.47 - 3.57 |
| + mooreScan - fusionLoss | 93.09 - 93.39 | 87.10 - 87.58 | 1.09 - 1.13 | 2.62 - 2.73 |
| - mooreScan + fusionLoss | 93.01 - 93.33 | 86.96 - 87.48 | 1.10 - 1.15 | 2.63 - 2.81 |
| + mooreScan + fusionLoss | 93.13 - 93.43 | 87.17 - 87.65 | 1.09 - 1.13 | 2.62 - 2.74 |
camus
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss | 92.12 - 93.20 | 85.40 - 87.24 | 1.89 - 1.98 | 3.69 - 3.94 |
| + mooreScan - fusionLoss | 93.67 - 94.65 | 88.12 - 89.80 | 1.29 - 1.38 | 2.75 - 3.02 |
| - mooreScan + fusionLoss | 93.40 - 94.46 | 87.65 - 89.45 | 1.27 - 1.36 | 2.84 - 3.14 |
| + mooreScan + fusionLoss | 93.77 - 94.83 | 88.31 - 90.11 | 1.31 - 1.40 | 2.78 - 3.08 |
echo-ped
| 扫描方式 | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| - mooreScan - fusionLoss | 90.90 - 91.84 | 83.46 - 84.76 | 1.65 - 1.85 | 4.21 - 4.78 |
| + mooreScan - fusionLoss | 91.75 - 92.61 | 84.88 - 86.12 | 1.56 - 1.73 | 3.73 - 4.18 |
| - mooreScan + fusionLoss | 91.88 - 92.74 | 85.10 - 86.34 | 1.53 - 1.70 | 3.70 - 4.16 |
| + mooreScan + fusionLoss | 91.98 - 92.88 | 85.28 - 86.58 | 1.52 - 1.66 | 3.65 - 4.18 |
5.5 WFL 组件消融实验
focal loss 的参数取值
γ (gamma): 默认值为2.0。这是smp.losses.FocalLoss的默认参数。
αt (alpha): 默认值为0.25。这也是smp.losses.FocalLoss的默认参数
| 扫描方式 | dsc | iou | ASSD | HD95 |
|---|---|---|---|---|
| dice v373 | 0.9266 | 0.8632 | 1.9351 | 3.8131 |
| jaccard v409 | 0.9292 | 0.8677 | 1.6377 | 3.6420 |
| focal v410 | 0.9282 | 0.8661 | 1.5135 | 3.4023 |
| dice + jaccard v411 | 0.9298 | 0.8688 | 1.6545 | 3.5315 |
| dice + focal v415 | 0.9347 | 0.8775 | 1.4783 | 3.2643 |
| jaccard+focal v413 | 0.9357 | 0.8793 | 1.4707 | 3.2732 |
| dice+jaccard+focal v278 | 0.9393 | 0.8855 | 1.3134 | 2.9897 |
测试最佳比例
9421 dice jaccard focal 0.4692 0.1687 0.3620
6. 对比实验
不同曲线的扫描
6.1 echo-Pediatric
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| 横线 | 91.37 | 84.11 | 1.75 | 4.50 |
| 竖线 | 90.61 | 82.83 | 1.76 | 4.31 |
| 斜线 | 91.21 | 83.84 | 1.65 | 4.17 |
| 希尔伯特 | 92.22 | 85.56 | 1.62 | 3.99 |
| Z阶 | 91.89 | 85.00 | 1.90 | 4.83 |
| moore | 92.43 | 85.93 | 1.59 | 3.91 |
横线
| Scan | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v242 | 0.9145 | 0.8425 | 1.5811 | 3.9611 | 0.9178 |
| v245 | 0.9137 | 0.8411 | 1.75 | 4.50 | |
| v355 | 0.9052 | 0.8268 | 1.7902 | 4.4417 |
竖线
| Scan | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v288 | 0.9149 | 0.8432 | 1.5771 | 3.9424 | 0.9224 |
| v292 | 0.9138 | 0.8413 | 1.6080 | 3.9769 | 0.9190 |
| v365 | 0.9061 | 0.8283 | 1.7608 | 4.3131 | 0.9284 |
斜线
| Scan | dsc | iou | assd | hd95 | sen |
|---|---|---|---|---|---|
| v290 | 0.9149 | 0.8431 | 1.5956 | 3.9276 | 0.9250 |
| v298 | 0.9135 | 0.8408 | 1.6367 | 4.1488 | 0.9254 |
| v302 | 0.9121 | 0.8384 | 1.6534 | 4.1673 | 0.9204 |
| v303 | 0.9145 | 0.8425 | 1.5952 | 3.9662 | 0.9183 |
hilbert
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V390 | 0.9222 | 0.8556 | 1.6232 | 3.9901 |
z-order
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V391 | 0.9220 3e4 | 0.8554 | ||
| V392 | 0.8246 1e5 | 0.7015 | ||
| V394 | 0.9189 | 0.8500 | 1.9004 | 4.8258 |
6.2 echo-dynamic
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| 横线 | 92.13 | 85.42 | 1.30 | 3.52 |
| 竖线 | 91.86 | 84.95 | 1.17 | 2.81 |
| 斜线 | 91.79 | 84.82 | 1.15 | 2.78 |
| 希尔伯特 | 91.97 | 85.13 | 1.17 | 2.83 |
| Z阶 | 93.11 | 87.12 | 1.14 | 2.74 |
| moore | 93.28 | 87.41 | 1.11 | 2.68 |
横线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V366 | 0.9213 | 0.8542 | 1.2950 | 3.5227 |
竖线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V382 | 0.9186 | 0.8495 | 1.1692 | 2.8081 |
斜线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V384 | 0.9179 | 0.8482 | 1.152 | 2.7794 |
hilbert
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V405 | 0.9180 | 0.8485 | ||
| V406 | 0.9197 | 0.8513 | 1.1674 | 2.8261 |
z-order
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V393 | 0.9311 | 0.8712 | 1.138 | 2.7374 |
6.3 camus
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| 横线 | 92.66 | 86.32 | 1.94 | 3.81 |
| 竖线 | 92.98 | 86.88 | 1.57 | 3.48 |
| 斜线 | 93.45 | 87.71 | 1.79 | 4.99 |
| 希尔伯特 | 93.43 | 87.67 | 1.55 | 3.41 |
| Z阶 | 93.15 | 87.18 | 1.52 | 3.52 |
| moore | 94.30 | 89.21 | 1.36 | 2.93 |
横线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| v373 | 0.9266 | 0.8632 | 1.9351 | 3.8131 |
竖线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V385 | 0.9298 | 0.8688 | 1.5653 | 3.4846 |
斜线
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V386 | 0.9345 | 0.8771 | 1.7853 | 4.9893 |
hilbert
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V387 | 0.9343 | 0.8767 | 1.5482 | 3.4110 |
| V388 | 0.9335 | 0.8753 |
z-order
| Scan | dsc | iou | assd | hd95 |
|---|---|---|---|---|
| V389 | 0.9315 | 0.8718 | 1.5158 | 3.5174 |
7. 修改意见
AE(执行编辑):审稿人对本文进行了详细评估,其意见如下。根据审稿人的反馈和我本人对稿件的评估,本文应仔细修改。作者应确保方法细节、实验设置等得到清晰阐明。请针对审稿人的意见提供逐点说明。请注意,最终录用取决于您修改的质量以及审稿人对您修改的完全满意。
审稿人 #1:
总结:该稿件目前形式存在方法缺乏清晰度、实验验证不足以及声称未得到充分证实等显著问题。
主要问题:
- 方法清晰度和可复现性不足:
- 图 2 模糊不清:图 2(a)、2(b) 和 2© 之间的关系不明确。图 2© 是描绘了两个连续的编码器层,还是 CASS 模块本身的详细示意图?
- 缺失架构细节:关键组件未定义。"Stem"模块未描述。此外,未说明解码器中"跳跃连接"是如何实现的(例如,逐元素相加、拼接或任何其他融合方法)。
- CASS/SSM 集成不清晰:图 2© 尤其令人困惑,因为它似乎只显示了扫描操作,而没有明确集成状态空间模型块,而这是基于 Mamba 的架构的基础。
- 强烈建议作者修改所有方法图和描述以提高清晰度,并公开其代码以确保可复现性。
- 论文指出摩尔曲线阶数 是基于特征图大小 "自动"选择的。然而,这种选择机制没有明确定义。公式 (5) 和 (6) 描述了曲线的属性,但没有提供用于在不同编码器层级选择 的显式映射函数或规则。
- WFL 模块的消融实验和细节不足:
- 缺失超参数:公式 (7) 中损失分量的权重 从未指定。也缺少关于不同权重分布如何影响分割性能的分析。
- 缺失组件消融实验:缺少一个关键的消融研究。作者应消融 WFL 的各个组件(例如,基线 vs. +Jaccard vs. +Dice vs. +Focal)以证明所有三个组件的必要性。
- 基线损失函数不明确:对于 M1 实验(无 WFL),论文没有说明使用了哪种损失函数作为替代。
- 实验对比不完整:
- 缺失扫描策略比较:核心声称是摩尔曲线的优越性。这应该与其他常见的空间填充曲线扫描方法进行基准测试,最著名的是希尔伯特曲线。
- 相关工作空白:相关工作部分未能讨论其他引入了替代扫描策略的基于 Mamba 的模型,这将是最相关的基线比较。
- 统计显著性:许多报告的性能提升是微小的。作者必须进行统计显著性检验(例如,报告配对 t 检验的 p 值),特别是对于"我们的方法"与"前三名 SOTA 方法"以及"我们的方法"与 M1/M2 的关键比较,以确认改进具有统计相关性。
- 效率声称无充分依据:作者声称"模型效率和实时处理",但仅报告了参数量。这是不够的。需要进行计算效率的综合比较,包括 GFLOPs、推理时间和训练时间,并与 SOTA 方法一起在表格中呈现。
- 可视化有限:提供的可视化(图 4, 6)仅显示了消融实验结果。为了证实优越性的声称,论文必须包括所提方法与对比 SOTA 方法在所有三个数据集的具有挑战性的案例上的定性视觉比较。建议使用高对比度的轮廓线。(未新增可视化图)
次要问题:
- 消融描述中的矛盾:第 4.4 节中存在明显矛盾。文本描述 M2 为"在 M1 中保留 WFL 模块,同时移除 CASS…"。然而,表 3 显示 M1 具有 CASS 而没有 WFL。这一定是文本中的笔误,需要更正。
- 注意力可视化模糊:在图 5 中,"注意力机制"热图的来源模糊不清。作者应说明此可视化源自网络的哪个部分(例如,哪一层,哪种注意力)。此外,应叠加真实分割掩码作为 ROI 的参考。****
- 缺失实现细节:
- 张量 的输入维度 D 在数据集预处理或方法部分从未指定。
- 图 2© 的逻辑非常令人困惑。它在视觉上暗示两个"有噪声的"特征图相结合产生了一个干净的 ROI,这是不直观的,需要明确的解释。
审稿人 #2:
本文提出了 EchoMamba,一种基于摩尔曲线引导的三维状态空间模型,用于超声心动图视频中的左心室分割,具有显著的创新性,但也存在需要修改的关键空白。
优点:
- 曲线自适应选择性扫描模块将摩尔曲线与 Mamba 集成,有效解决了传统线性扫描的端-始不连续性问题,与心动周期的时空周期性相吻合。多阶曲线映射平衡了全局解剖拓扑和局部边缘特征,缓解了超声图像各向异性分辨率的问题。
- 加权融合损失结合了 Jaccard、Dice 和 Focal 损失,针对超声心动图中类别不平衡和边界模糊的关键痛点。
- 在三个数据集上的实验表明,相较于 SOTA 方法,在 Dice、IoU 和边界指标上表现更优,同时 24.1M 的参数量保持了轻量级潜力,利于临床部署。
主要关切:
- 方法清晰度与可复现性
WFL 的权重仅在数学上定义但未说明理由:是凭经验选择的吗?没有消融或对比实验验证最优值,削弱了损失函数的严谨性。
摩尔曲线生成(例如,坐标转换到 [0, L-1]×[0, L-1])和曲线阶数选择(3-6)缺乏原理说明。CASS 与 Mamba 状态更新之间的交互仍不清楚。 - 实验设计缺陷
表 2 中,参数量错误地将"我们的方法"加粗,尽管 24.1M 并非最小(例如,UKAN3D:9.1M,SegFormer3D:7.5M)。表 3 也对非最优数据加粗(例如,CAMUS 的 ASSD 中"我们的方法"是 1.36,而 M2 的是 1.31),误导读者——必须更正格式。
60 个训练周期的充分性未得到验证:没有提供收敛曲线或分析来确认模型达到了稳定性能,可能危及结果的可靠性。 - 其他
注意力热图模糊;隐藏状态演化缺乏解剖结构映射,降低了临床可信度。(待补充图)
修改建议:
- 更正表 2 和表 3 中的加粗,严格与最优性能对齐:仅对每个指标在所有对比方法中排名第一的值加粗(例如,在表 2 中,移除"我们的方法"在"参数量"下的加粗,因为它不是最小的;在表 3 中,调整如 CAMUS ASSD 等指标的加粗以突出表现最佳的条目)。
- 验证 60 个训练周期的充分性:要么提供收敛曲线(例如,训练/验证损失、Dice 系数随周期的变化趋势)以确认模型在 60 个周期达到了稳定性能,要么进行更多周期(例如 200 或更多)的补充实验以排除不完全收敛对结果的影响。
- 将扫描策略消融研究(比较摩尔、扫描、垂直和对角线扫描)扩展到所有三个数据集,而不仅限于 EchoNet-Peds。这将验证摩尔曲线扫描在不同数据分布下的普适性。
- 提高可视化图的清晰度(例如,图 4, 5, 6):增加图像分辨率以确保分割轮廓、注意力热图区域以及扫描策略之间的差异等细节可辨。
审稿人 #3:
该稿件提出了一种基于摩尔空间填充曲线引导的状态空间方法,用于超声心动图视频中的左心室分割。该方法结合了曲线自适应选择性扫描(闭环摩尔扫描)、跨编码器尺度的多阶映射策略以及加权损失(Jaccard + Dice + Focal)。在 EchoNet-Dynamic、CAMUS 和 EchoNet-Peds 上的实验报告了有竞争力的数值,并显示摩尔扫描优于几种简单替代方案。该方向很有前景,但在方法规范、基线的公平性、临床相关性、泛化性、效率报告和可复现性方面存在重大空白。我建议重大修改。此外,请考虑重命名该方法:"EchoMamba"这一名称已在其他已发表工作中出现,可能会在索引和引用中引起混淆。
- 基线套件遗漏了几个 2024-2025 年强大且广泛采用的比较器。除了已引用的 CNN/Transformer/扩散和 SAM 衍生的基线外,请包括:(i) 具有可靠时空记忆提示的 MemSAM;(ii) MedSAM 或类似的高容量基础模型作为通用基线;(iii) 近期基于 Mamba/SSM 的医学分割模型(例如,SegMamba 变体)。匹配训练预算和硬件,并报告精度-速度-内存的权衡。没有这些,增量价值难以判断。
- 论文解释了空间摩尔映射,但没有说明用于形成 SSM 输入的时空交织方式:特征是逐帧扫描然后跨时间拼接、跨帧交织,还是以滑动窗口方式打包;循环状态如何跨帧传递;以及如何处理重置/填充;还有训练和推理中使用的窗口长度。
- Jaccard、Dice 和 Focal 分量的权重,以及 Focal 参数(α, γ),未按数据集报告,也未说明其选择理由。
- 声称优于 SAM 风格和扩散基线的说法需要更强的公平性文档。对于每个比较器,请指定提示协议(点/框,记忆长度,关键帧策略)、微调程度、数据增强和计算预算对等性。对于非提示模型(例如 UKAN3D 或 Transformer 基线),记录推荐的超参数、早停标准和模型选择标准。没有这些,比较可能有利于所提出的方法。
- 核心假设——闭环扫描改善了 ES↔ED 边界稳定性和周期性一致性——主要得到通用分割指标和定性可视化的支持。请添加相位条件指标(例如,按心脏相位分箱的 Dice/HD95)和临床有意义的结果,如 EDV/ESV/EF 误差和相关性,以证明切实的临床益处。
- 包括跨数据集/设备迁移(例如,在 EchoNet-Dynamic 上训练 → 在 CAMUS 上测试),按供应商/帧率分层,并评估非周期性模式(例如,心律失常)或提供代理分析。报告关键增益的置信区间和统计显著性。讨论部分承认对非周期性运动的适应性有限——请量化这一局限性。
- 报告吞吐量(fps)、每序列延迟、内存使用情况和 FLOPs/MACs,最好有设备上的测量结果(例如,笔记本/移动 GPU)。在相同硬件和数值精度下,绘制与代表性 Transformer/Mamba/SAM 基线的精度-速度边界图。这对于旨在实时或近实时使用的视频分割系统至关重要。
- 定义所有常数(例如,公式中的 ε),并阐明多阶映射中的舍入/边界规则。关于摩尔路径是"可学习的"的陈述是模糊的,因为摩尔曲线是确定性的:请澄清实际学习的是什么(例如,阶数选择、门控或参数化遍历)。记录完整的预处理/后处理(裁剪、归一化、时间采样)、确切的数据分割和训练计划。发布代码/配置和训练好的权重以便验证。
- 加强实验设计以反映当前文献并相应调整框架:(a) 添加具有时序记忆提示的 MemSAM;(b) 包括 MedSAM(如果可行,还包括嵌入式/高效变体)以说明临床可部署性;© 添加 SegMamba 或密切相关的 SSM 基线作为代表。一些医学领域模型,如 VM-UNet / Mamba-UNet/ LoG-VMamba/ LKM-UNet/H-vmunet, nnunet。
总结
| 主要方向 | 核心修改意见 | 建议的解决措施 |
|---|---|---|
| 方法细节 | 审稿人1, 2, 3均指出: 方法描述模糊,关键细节缺失(如Stem模块、跳跃连接方式、CASS与Mamba的集成、多阶摩尔曲线选择机制、WFL超参数、时空处理逻辑),导致难以理解和复现。 | 1. 彻底重写方法章节:用更清晰的文字和重新绘制的图表(如Fig.2)详细解释每个模块。 2. 补充所有缺失细节:明确说明架构设计、超参数值(如WFL的x,y,z)、摩尔曲线阶数k的选择规则、输入张量维度D等。 3. 公开代码和模型:在修订稿中承诺并在录用后立即公开代码和配置,这是最有力的回应。 |
| 增加实验 | 审稿人1, 3均指出: - 基线对比不足,缺少与近期SOTA方法的对比。 - 扫描策略对比不完整(缺少与Hilbert曲线等的比较)。- WFL等组件的必要性未通过充分的消融实验证明。 - 效率声称不完整(仅参数量,缺FLOPs、推理时间)。 | 1. 增补sota实验:加入审稿人3建议的多个最新基线模型(如 Mamba-UNet,LKM-UNet,SegMamba,其他引入了替代扫描策略的mamba),并在相同条件下进行公平比较。 2. 扩展对比实验:将摩尔曲线与希尔伯特曲线等更多空间填充扫描策略进行对比,并在所有三个数据集上进行验证。 3. WFL的组件消融实验 :增加对WFL各损失分量的逐项消融,证明其组合的必要性 4. 补充效率数据:在结果表格中增加GFLOPs、推理速度(FPS)和内存消耗等指标,全面评估效率。 |
| 实验细节 | 审稿人1, 2, 3均指出: - 性能提升的统计显著性未验证。 - 训练周期(60 epochs)是否足够未验证。- 缺乏临床指标(如EF评估误差)来证明实际价值。 | 1. 进行统计检验:对关键对比结果(如vs. SOTA, vs. M1/M2)进行统计显著性检验(如p值),并在文中报告。 2. 证明训练充分性:在补充材料中提供训练/验证集的损失和Dice系数收敛曲线,证明60 epochs已足够。3. 补充临床指标:计算并报告左心室容积(EDV, ESV)和射血分数(EF)的误差与相关性,强化学术价值。 |
| 可视化 | 审稿人1, 2, 3均指出: - 定性对比不足,缺少与SOTA方法的可视化效果图。 - 现有可视化(如注意力热图)模糊,缺乏解释。 | 1. 增加定性对比:提供所提方法与主要SOTA基线在挑战性案例上的分割效果对比图,用高对比度轮廓清晰展示优势。 2. 优化并解释可视化:提高图片分辨率,清晰说明注意力热图源自网络的哪个部分,并叠加GT轮廓作为参考。 |
| 格式 | 审稿人1, 2, 3均指出: - 表格中的加粗格式错误(对非最优结果加粗)。 - 文本中存在矛盾描述(如M1/M2的配置)。 - 方法名称“EchoMamba”可能已被占用。 | 1. 彻底检查并修正:仔细检查所有表格,确保只对性能最优的指标加粗。 2. 统一并更正文本:修正关于M1/M2描述的矛盾之处,确保全文表述一致。 3. 考虑重命名:认真考虑审稿人3的建议,为方法寻找一个更具独特性的新名称,以避免混淆。 |